Article
국내 유통 목재 펠릿 구성 수종 확인을 위한 메타바코딩 분석
이병주1,
안지영2,
어수형1,*
Metabarcoding Profiling of Wood Pellet Domestically Supplied in South Korea
Byoeng-Ju Lee1,
Ji Young Ahn2,
Soo Hyung Eo1,*
Author Information & Copyright ▼
1Department of Forest Science, Kongju National University, Yesan 32439, Korea
2Division of Forest Bioinformation, National Institute of Forest Science, Suwon 16631, Korea
© Copyright 2024 Korean Society of Forest Science. This is an Open-Access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/4.0/) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Received: Apr 19, 2024; Revised: Aug 02, 2024; Accepted: Nov 06, 2024
Published Online: Dec 31, 2024
요 약
전세계적으로 불법 목재 방지 및 합법 목재 유통을 촉진하기 위한 합법목재교역촉진제도가 시행되고 있으며, 국내에서도 관련법을 통해 수입 목제 제품에 대한 합법성을 검증하고 있다. 목재 펠릿과 같은 복수 수종 구성 제품의 경우, 입증자료 내 구성 수종이 현물과 일치하는지 확인하기 위한 과학적인 검증 방법이 필요하다. 본 연구에서는 ITS2 영역 메타바코딩 분석법을 통한 목재 펠릿 구성 수종 동정 방법을 제시하고, 분석 결과를 통한 유의사항을 살펴보고자 한다. 분석 결과, 총 40개의 ASV(Amplicon Sequence Variant)가 형성되었으며, 35개 ASV가 속(genus) 수준까지 동정되었다. 35개 중 3개 ASV는 근연종으로 분류되었으며, 2개 ASV는 식물계(Plantae)에 포함되지 않는 것으로 확인되었다. 목재 펠릿 구성 수종으로는 Pinus 속, Larix 속, Quercus 속, Rosa 속 등이 확인되었으며, 산림에 주로 분포하는 분류군이 포함된 것으로 판단된다. 향후 메타바코딩 분석 방법의 구성 수종 식별 정확성을 측정하기 위해 전체 구성 확인이 가능한 시료를 활용하여 추가적인 연구가 필요할 것이다. 또한, 일부 근연종에 대한 분류 능력을 높이고, 대상 외의 분류군을 걸러내기 위한 방법으로 chloroplast DNA 영역에 대한 메타바코딩 분석 또한 필요할 것으로 판단된다.
Abstract
A legal wood trade promotion system has been implemented globally to prevent illegal wood trading and promote the supply of legal wood. In Korea, laws require the verification of the legality of imported wood products. For products composed of multiple species, such as wood pellets, a verification method is necessary to confirm that the species listed in the submitted data correspond to the actual product. In this study, we propose ITS2 region metabarcoding analysis for identifying the species composition of wood pellets and discuss key considerations based on the results. As a result of the analysis, 40 amplicon sequence variants (ASVs) were clustered, and 35 ASVs were identified at the genus level. Among the 35 ASVs, 3 ASVs were identified as closely related multiple species, and 2 ASVs were identified as taxa not belonging to Plantae. The wood pellets included genera such as Pinus, Larix, Quercus, and Rosa, suggesting mostly forest species. In the future, further research is needed to assess the accuracy of taxa identification using metabarcoding methods, employing wood pellet samples with a fully known composition. Additionally, metabarcoding analysis of the chloroplast DNA region may be necessary to improve the discriminatory ability for closely related species and to filter out nontarget taxa.
Keywords: metagenomics; high throughput sequencing; tree species OTU; wood forensic
서 론
전세계 불법 목재 유통은 15~30%으로 예상되며(Nellemann, 2012), 불법 목재 생산을 위한 불법 벌채는 산림 파괴에 의한 생물다양성 감소와 기후 온난화 등에 문제를 야기하며, 목재 시장을 교란한다(Reboredo, 2013; Dormontt et al., 2015),. 우리나라는 목재 자급률이 2022년 기준 15%로 수입 의존도가 높아, 불법 목재 유통에 대한 우려가 높은 상황이다(Ahn, 2024).
APEC(Asia Pacific Economic Cooperation)에서는 회원국들에게 합법적 목재 교역 촉진을 위한 제도 도입을 권장하고 있으며, 미국, 호주, 일본 등을 비롯한 회원국들에서는 관련 법률을 제정하여 시행 중이며, 국내에서는 ‘목재의 지속가능한 이용에 관한 법률’을 개정하여 2018년도부터 합법목재 교역촉진제도를 시행하고 있다(Ahn, 2024). 해당 법률에서는 원목, 제제목, 합판, 목재 펠릿 등 수입 목재 제품을 대상으로 수입업자가 합법성을 입증하도록 하고 있다. 특히, 합판이나 목재 펠릿과 같은 혼합 목재제품의 합법성 입증은 제품의 목재수종 비율표, 원산지, 원자재별 공급업체 정보 등 자료를 제출하도록 되어있다. 따라서, 업자의 제출 자료 내 구성 수종 정보와 현물의 일치 여부를 확인하기 위한 과학적 식별 기술이 필요하다(Ahn, 2024).
기존에는 현미경을 사용한 해부학적 분류에 의존하여 목재 수종 식별이 이루어져 왔으나(Dormontt et al., 2015), 종 수준의 분류가 불가능하고(Gasson, 2011), 해부학적 지식이 있는 전문가가 필요하다는 단점이 있다(Dormontt et al., 2015). DNA 분석 기법은 생물 유전체의 일부 염기서열 정보로 종, 개체 등의 정보를 추적하는 방법이다(Dormontt et al., 2015; Jiao et al., 2020). 수종 식별에는 주로 matK, rbcL, psbC 등 chloroplast DNA 영역이나, ITS와 같은 nuclear DNA의 염기서열 정보가 사용된다(Cheng et al., 2016; Jiao et al., 2020). 이중 ITS 영역은 종간 식별 능력이 높아, 식물 종 분류에 널리 사용되고 있다(Cheng et al., 2016).
메타바코딩 분석법은 기존의 단일 종 DNA 염기서열을 분석하는 것과는 달리, 여러 종이 섞여 있는 시료의 유전체를 분석하는 데 사용되는 기법이다(Deiner et al., 2017). 물, 토양, 분변 등 복수 종이 섞여 있는 시료 유전체를 대상으로 HTS(High Throughput Sequencing) 분석을 실시하여 단시간 내에 대량의 유전 정보를 얻어내 종 식별에 사용할 수 있다(Taberlet et al., 2012; Creer et al., 2016; Denier et al., 2017). 목재 펠릿과 같이 여러 수종이 섞여 있는 목재 제품을 대상으로 메타바코딩 분석법이 적합할 것으로 판단된다.
본 연구에서는 복수 수종 목재 제품의 구성 수종 식별 기술로써 국내 유통 목재 펠릿으로부터의 DNA 추출부터 ITS2 영역 메타바코딩 분석, BLAST 수종 동정까지의 방법론을 제공하고자 한다. 또한 연구가 거의 이루어지지 않았던 목재 펠릿 유전체 메타바코딩 분석에 대한 사전 연구로써 필요 사항 및 주의점을 파악하여 제시하고자 한다.
재료 및 방법
1. 목재 펠릿 파쇄
국내에서 생산되는 목재 펠릿 제품을 구매하여 연구에 사용하였다. 목재 펠릿 DNA 추출에 앞서, 멸균된 망치를 이용하여 파쇄하였다. 파쇄 시에는 테이블에 멸균된 알루미늄을 호일을 깔았으며, 매 파쇄시 마다 호일을 새 것으로 교체하고 망치 또한 1차 증류수, 차아염소산나트륨(10%), 알코올(70%), 1차 증류수 순서로 세척하여 사용하였다. 9개 목재 펠릿을 파쇄하였으며, 파쇄된 시료는 펠릿별로 다른 conical tube에 옮겨 담아 DNA 추출용 재료로 사용하였다. 파쇄 후 시료의 오염 및 변형을 방지하기 위해, 지체없이 다음 실험을 진행하였다.
2. 목재 펠릿 DNA 추출
파쇄된 시료 0.2g씩 분주하여 DNA 추출에 사용하였다. DNA 추출에는 Qiagen DNeasy Plant Maxi kit (Qiagen Hilden, Germany)를 사용하였으며, 실험 방법은 DNeasy Plant Handbook의 “purification of Total DNA from Plant Tissue (Maxi Protocol)”을 따라 진행되었다. 모든 과정의 원심분리는 4,255 xg에서 실시하였고, 최종 dilution 과정의 AE buffer는 750uL를 사용하였다.
추출된 DNA 시료의 농도를 높이기 위해 10배 농축을 실시하였다. 농축 과정은 기존의 에탄올 침전법(Green and Sambrook, 2016)을 따랐으며, 과정은 다음과 같다. 700uL DNA 용액에 Sodium acetate(3M)가 최종 용액 농도 0.3M이 되도록 77.8uL을 추가하였다. 이후 Linear polyacrylamide (5mg/mL)를 최종 용액 내 20ng/uL 농도가 되도록 3.1 uL를 첨가하였다. 0°C에 보관한 95% enthanol을 이전 단계 용액 양의 2배를 첨가한 뒤, 파쇄 얼음에 30분간 정치하여 인큐베이션하였다. 이후 0°C에서 15,000rpm으로 10분간 원심분리 실시 후, pipet을 이용하여 침전물을 건드리지 않으며 상층액을 전부 제거하였다. 침전물만 남은 튜브에 70% ethanol 750uL을 첨가한 후 4°C에서 15,000rpm으로 2분간 원심분리 실시 후, pipet을 이용해 침전물을 건드리지 않고 상층액을 전부 제거하였다. 침전물만 남은 튜브의 뚜껑을 열어 상온에 1시간 정치하여 잔여 ethanol을 증발시켜 제거한 후, TE 용액 70uL를 추가하고 침전물이 완전히 녹도록 튜브를 가볍게 뒤집으며 흔들어 주었다. 농축된 DNA는 다음 실험 전까지 −20°C에 냉동 보관하였다.
3. ITS2 영역 metabarcoding 분석
메타바코딩 분석을 위한 ITS2 영역(Table 1) 라이브러리 제작은 Illumina 16s rRNA metagenomic sequencing library protocol을 참고하여 이루어졌다. 1차 PCR은 gDNA 10ng, reaction buffer 5X, dNTP 1mM, forward/reverse primer 각각 500nM, Herculase II fusion DNA polymerase (Agilent Technologies, Santa Clara, USA)를 혼합하여 반응액을 제조하였다. 1차 PCR의 cycle은 94°C에서 4분간 initial denaturation 후, denaturation을 94°C에서 30초, annealing을 55°C에서 40초, extension을 72°C에서 60초로 34회 반복하였으며, 72°C에서 10분간 final extension을 실시하였다. 1차 PCR에 사용된 Illumina adapter 부착 ITS2 universal primer의 염기서열은 다음과 같다. ITS3p forward primer 5’ TCGTCGGCAGCGTCAGATGTGTATAAGAG ACAGYGACTCTCGGCAACGGATA 3’, ITS4u reverse primer 5’ TCGTGGGCTCGGAGATGTGTATAAGAGAC AGRGTTTCTTTTCCTCCGCTTA 3’. 1차 PCR 산물은 AMPure beads (Agencourt Bioscience, Beverly, USA)로 purification 하였다. 2차 Index PCR은 Nextera XT Index primer를 사용하였으며, 증폭 조건은 1차 PCR 조건과 동일하였다. 2차 PCR 산물 또한 AMPure baeds를 사용하여 purification 하였다. 제작된 library를 대상으로 MIseq 300PE platform(Illumina, San Diego, USA)을 통해 염기서열 분석을 실시하였다. 라이브러리 제작과 염기서열 분석은 (주) MACROGEN에 의뢰하여 이루어졌다.
Table 1.
Primer set used for metabarcoding.
| Primer |
Sequence(5’→3’) |
Direction |
Reference |
| ITS-3p |
YGACTCTCGGCAACGGATA |
Forward |
Cheng et al., 2016
|
| ITS-4u |
RGTTTCTTTTCCTCCGCTTA |
Reverse |
Download Excel Table
분석된 염기서열은 QIIME2-cutadapt(--p-front-f YGACT CTCGGCAACGGATA, --p-front-r RGTTTCTTTTCCTCC GCTTA)를 통해 염기서열의 primer 영역을 제거하였다. 이후 QIIME2-DADA2(--p-trunc-len-f 0, --p-trunc-len-r 0)를 통해 quality filtering, denoising, dereplication과 동시에 ASV(Amplicon Sequence Variant)를 형성하였다. ASV의 염기서열을 대상으로 NCBI nt database를 사용하여 BLAST(Geneious 10.2.5)를 실시하였다. BLAST 결과 중 E-value가 가장 낮고, identical score와 query coverage가 80% 이상인 것을 동정 결과로 선택하였으며, 2개 이상의 종에 대해 BLAST 결과(e-value, identical score, query coverage)가 동일한 경우 복수 종이 동정된 것으로 표시하였다.
결과 및 고찰
염기서열 분석결과 3,262,500개, 총 길이 982,012,500 bp의 염기서열이 분석되었다. Quality filtering 및 denoising 후 총 8,506개 reads가 ASVs 형성에 사용되었으며, 총 40개의 ASVs가 만들어졌다(Table 2).
Table 2.
The number of reads of ASV in each wood pellet sample.
| ASV_ID |
P01 |
P02 |
P03 |
P04 |
P05 |
P06 |
P07 |
P08 |
P9 |
| ASV001 |
0 |
0 |
0 |
0 |
0 |
0 |
850 |
0 |
0 |
| ASV002 |
774 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
| ASV003 |
0 |
0 |
0 |
0 |
0 |
0 |
762 |
0 |
0 |
| ASV004 |
0 |
0 |
0 |
651 |
0 |
0 |
0 |
0 |
0 |
| ASV005 |
0 |
0 |
0 |
457 |
0 |
0 |
0 |
0 |
0 |
| ASV006 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
417 |
| ASV007 |
0 |
371 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
| ASV008 |
0 |
0 |
0 |
0 |
353 |
0 |
0 |
0 |
0 |
| ASV009 |
0 |
0 |
0 |
0 |
0 |
0 |
310 |
0 |
0 |
| ASV010 |
0 |
0 |
298 |
0 |
0 |
0 |
0 |
0 |
0 |
| ASV011 |
0 |
0 |
0 |
294 |
0 |
0 |
0 |
0 |
0 |
| ASV012 |
0 |
0 |
0 |
0 |
291 |
0 |
0 |
0 |
0 |
| ASV013 |
0 |
275 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
| ASV014 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
260 |
0 |
| ASV015 |
190 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
| ASV016 |
179 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
| ASV017 |
0 |
0 |
0 |
0 |
162 |
0 |
0 |
0 |
0 |
| ASV018 |
0 |
159 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
| ASV019 |
0 |
146 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
| ASV020 |
0 |
0 |
132 |
0 |
0 |
0 |
0 |
0 |
0 |
| ASV021 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
130 |
0 |
| ASV022 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
129 |
0 |
| ASV023 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
127 |
0 |
| ASV024 |
0 |
0 |
0 |
81 |
0 |
0 |
0 |
0 |
0 |
| ASV025 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
79 |
0 |
| ASV026 |
0 |
0 |
0 |
0 |
78 |
0 |
0 |
0 |
0 |
| ASV027 |
73 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
| ASV028 |
0 |
70 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
| ASV029 |
64 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
| ASV030 |
0 |
64 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
| ASV031 |
0 |
0 |
0 |
0 |
59 |
0 |
0 |
0 |
0 |
| ASV032 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
40 |
0 |
| ASV033 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
35 |
| ASV034 |
0 |
0 |
0 |
0 |
31 |
0 |
0 |
0 |
0 |
| ASV035 |
28 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
| ASV036 |
27 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
| ASV037 |
0 |
0 |
0 |
0 |
25 |
0 |
0 |
0 |
0 |
| ASV038 |
0 |
0 |
0 |
0 |
0 |
0 |
17 |
0 |
0 |
| ASV039 |
0 |
0 |
0 |
0 |
0 |
0 |
15 |
0 |
0 |
| ASV040 |
0 |
0 |
0 |
0 |
0 |
3 |
0 |
0 |
0 |
Download Excel Table
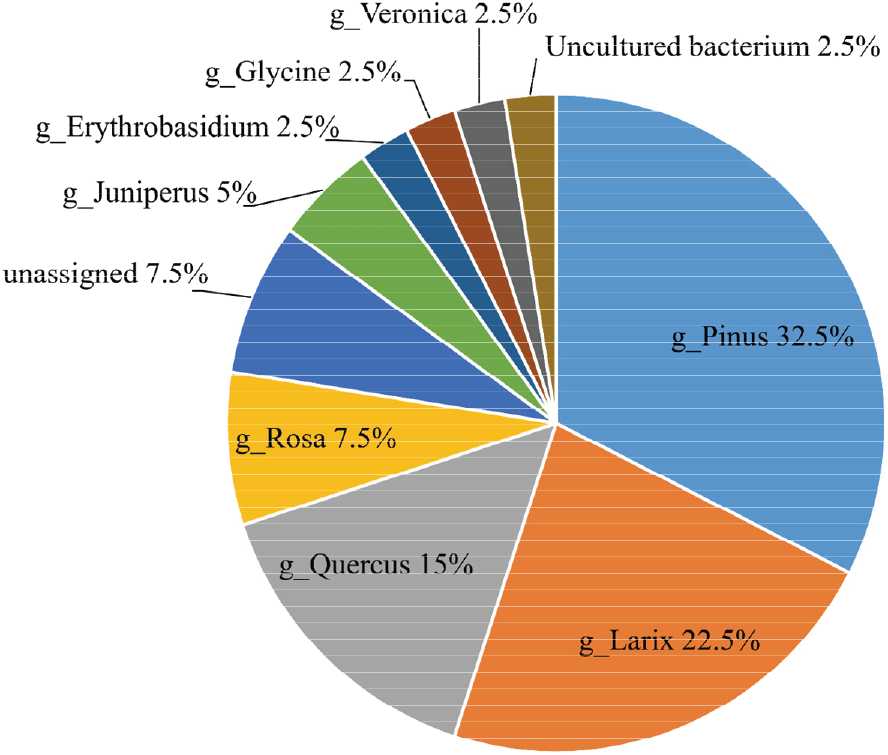
40개 ASV 중 35개가 동정에 성공하였으며, 분류된 속(genus)별 비율은 Pinus 32.5%, Larix 22.5%, Quercus 15%, Rosa 7.5% 등의 순서로 나타났다(Table 3, Figure 1). 해당 속들은 전세계적으로 분포 범위가 넓고, 경제적으로도 광범위하게 사용되는 분류군들이다(Gernandt et al., 2005; Bruneau et al., 2007; Goryachkina et al., 2013; Hubert et al., 2014). 속 수준 동정이 된 ASV 중, ASV005, ASV014, ASV018은 단일 종을 특정하는 데 실패하였다. ASV005가 분류된 Pinus sylvestris, P. thunbergii, P. taiwanensis, P. hwangshanensis은 Pinus 아절(subsection) 포함되어 유전적으로도 매우 가까우며(Zeb et al., 2020), ASV014(Quercus dentate, Q. mongolica, Q. serrata)와 ASV018(Quercus variabilis, Q. acutissima)이 분류된 종들은 Quercus 속내에서 Quercus 절(section)과 Cerris 절로 각각 나뉘며, 각 절 내 종들은 유전적으로 가까운 근연 관계이다(Zhou et al., 2022). 종간 매우 가까운 근연 관계에 의해 ITS 영역 염기서열이 동일하여, 종 수준에서 단일 종으로 분류가 실패한 것으로 판단된다.
Table 3.
ASV and taxa identified using BLAST(nr database) in wood pellet samples.
| ASV_ID |
Genus |
species |
E-value |
Identical score |
Query coverage |
| ASV001 |
(unassigned) |
(unassigned) |
- |
- |
- |
| ASV002 |
Rosa
|
Rosa soulieana
|
0 |
99.2 |
99.75 |
| ASV003 |
Quercus
|
Quercus serrata
|
0 |
99.2 |
100 |
| ASV004 |
(unassigned) |
(unassigned) |
|
|
|
| ASV005 |
Pinus
|
Pinus sylvestris; Pinus thunbergii; Pinus taiwanensis; Pinus hwangshanensis
|
0 |
96 |
100 |
| ASV006 |
Pinus
|
Pinus nigra
|
0 |
95.6 |
100 |
| ASV007 |
Quercus
|
Quercus aliena
|
0 |
99.5 |
99.75 |
| ASV008 |
Pinus
|
Pinus contorta
|
5.89E-165 |
92.2 |
98.13 |
| ASV009 |
Pinus
|
Pinus contorta
|
0 |
98.8 |
100 |
| ASV010 |
Larix
|
Larix decidua
|
0 |
97 |
99.76 |
| ASV011 |
Larix
|
Larix decidua
|
0 |
94.6 |
100 |
| ASV012 |
Juniperus
|
Juniperus communis
|
0 |
100 |
100 |
| ASV013 |
Pinus
|
Pinus contorta
|
1.31E-146 |
89.1 |
100 |
| ASV014 |
Quercus
|
Quercus dentata; Quercus mongolica; Quercus serrata
|
0 |
99.7 |
100 |
| ASV015 |
Larix
|
Larix decidua
|
0 |
95.1 |
100 |
| ASV016 |
Larix
|
Larix decidua
|
0 |
95.3 |
100 |
| ASV017 |
(unassigned) |
(unassigned) |
- |
- |
- |
| ASV018 |
Quercus
|
Quercus variabilis; Quercus acutissima
|
0 |
99.2 |
100 |
| ASV019 |
Pinus
|
Pinus contorta
|
0 |
95.1 |
100 |
| ASV020 |
Larix
|
Larix decidua
|
0 |
97.6 |
100 |
| ASV021 |
Pinus
|
Pinus contorta
|
2.15E-154 |
90.2 |
100 |
| ASV022 |
Pinus
|
Pinus nigra
|
1.23E-125 |
97.8 |
85.35 |
| ASV023 |
Pinus
|
Pinus contorta
|
0 |
94.6 |
100 |
| ASV024 |
Larix
|
Larix decidua
|
0 |
95.8 |
100 |
| ASV025 |
Larix
|
Larix decidua
|
0 |
97.3 |
100 |
| ASV026 |
(unassigned) |
(unassigned) |
- |
- |
- |
| ASV027 |
Rosa
|
Rosa soulieana
|
0 |
98.2 |
99.75 |
| ASV028 |
Erythrobasidium
|
Erythrobasidium sp.; Erythrobasidium hasegawianum |
0 |
99.5 |
100 |
| ASV029 |
Glycine
|
Glycine max
|
0 |
100 |
100 |
| ASV030 |
Quercus
|
Quercus aliena
|
0 |
97.7 |
99.75 |
| ASV031 |
Veronica
|
Veronica arvensis
|
0 |
99.5 |
98.28 |
| ASV032 |
(unidentified) |
Uncultured bacterium |
3.40E-161 |
99.4 |
98.16 |
| ASV033 |
(unassigned) |
(unassigned) |
- |
- |
- |
| ASV034 |
Pinus
|
Pinus contorta
|
9.65E-178 |
93.5 |
100 |
| ASV035 |
Larix
|
Larix decidua
|
0 |
96.3 |
100 |
| ASV036 |
Rosa
|
Rosa soulieana
|
0.00E+00 |
98.2 |
99.75 |
| ASV037 |
Juniperus
|
Juniperus communis
|
0 |
98.3 |
100 |
| ASV038 |
Quercus
|
Quercus serrata
|
0.00E+00 |
98.2 |
100 |
| ASV039 |
Pinus
|
Pinus nigra
|
2.73E-168 |
92.1 |
100 |
| ASV040 |
Ascidia
|
Ascidia ahodori
|
1.14E-145 |
98.3 |
100 |
Download Excel Table
동정된 분류군들 중 목재 수종 외의 종들도 포함되어 있었다(Table 3). 콩속(Glycine), 개불알풀속(Veronica)은 초본 식물 분류군으로, 목재의 벌채나 운송 과정에서 포함된 것으로 생각된다. Erythrobasidium 속은 효모(yeast)에 속하는 속으로, 기존 사례에서 식물 시료로부터 분리된 경우가 있었고(Nakase et al., 2003), 본 연구에서도 식물체에 서식하던 효모의 유전체가 분석에 포함된 것으로 판단된다. Ascidia 속은 멍게, 미더덕이 포함된 해초강(Ascidiacea)에 속하며, 바다에 서식하는 분류군이다(Shenkar and Swalla, 2011). 목재 자재의 운송 과정에서 바다모래와 같은 건축 자재의 운송 장비가 겸용되어 섞여 들어간 유전체인 것으로 여겨진다. 이와 같이 원자재 자체 또는 제작 및 운송 과정에서 포함되는 목재 수종 이외의 유전체가 나타날 수 있다는 점을 분석에 있어 고려해야 할 것이다.
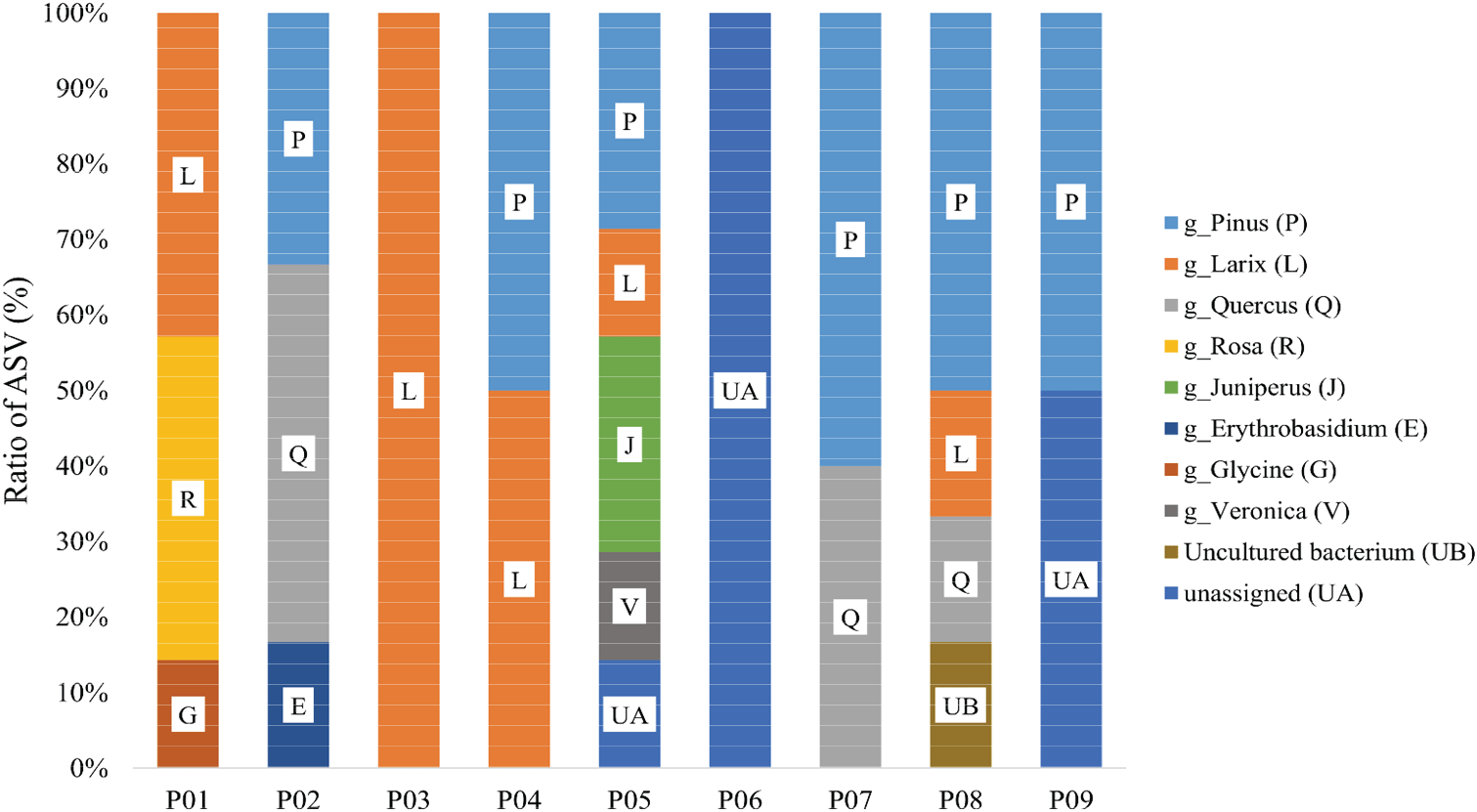
실험에 사용된 9개 시료들 각각의 구성 수종 동정 결과에서는 시료별 분류군 구성에 차이가 큰 것으로 나타났다(Figure 2). 제작 과정 중 파쇄된 목재가 고르게 배합되지 않기 때문으로 생각된다. 펠릿 제품의 복수 수종 동정에 있어 소수의 시료를 사용하는 것은 잘못된 동정 결과를 야기할 수 있으므로, 다수의 시료가 분석에 활용되어야 할 것이다.
본 연구는 시중에 판매되는 목재 펠릿 내 구성 수종 동정을 위한 메타바코딩 분석을 실시하고, 해당 방법론과 결과의 유의점을 제공하는데 의의가 있다고 판단된다. 다만, 구매 과정 중 정확한 구성 수종을 알 수 없어, 유전 분석 결과의 정확도를 파악할 수는 없었다. 추후 제작 과정부터 원자재 수종이 파악 가능한 제품이나 자체 제작한 펠릿 시료를 활용하여 메타바코딩 분석법의 정확도를 측정할 필요가 있으며, 이와 함께 기존 해부학적 방법과의 효율성 비교도 필요할 것으로 판단된다. 또한 일부 종 수준에서의 변별력이 떨어지는 문제(e.g. ASV005, ASV014, ASV018)와 식물 분류군 이외의 유전체가 포함되는 문제(e.g. Ascidia)를 해결하기 위해, 식물 분류군에 특이적으로 적용할 수 있는 chloroplast DNA 영역 메타바코딩 분석을 추가로 실시하여 ITS 영역과의 교차 검증으로 정확성을 높일 수 있을 것이다.
감a사의 글
본 연구는 국립산림과학원의 “산림생명자원 유전정보 구축 및 바이오지문 분석기술 개발 연구(과제 번호: FG06 01-2019-02)”의 지원으로 수행되었습니다. 또한, 시료 준비 및 DNA 추출 과정에 도움을 주었던 이택규 학생에게 감사의 마음을 표합니다.
References
Ahn, J.Y. 2024. International trends and Korea development status of timber species identification technology to comply with the legal timber trade promotion system. National Institute of Forest Science (NIFoS), Forest Policy Issue. pp. 15-16.

Bruneau, A., Starr, J.R. and Joly, S. 2007. Phylogenetic relationships in the genus
rosa: New evidence from chloroplast DNA sequences and an appraisal of current knowledge. Systematic Botany 32(2): 366-378.


Cheng, T., Xu, C., Lei, L., Li, C., Zhang, Y. and Zhou, S. 2016. Barcoding the kingdom plantae: New PCR primers for ITS regions of plants with improved universality and specificity. Molecular Ecology Resources 16(1): 138-149.



Creer, S., Deiner, K., Frey, S., Porazinska, D., Taberlet, P., Thomas, W.K., Potter, C. and Bik, H.M. 2016. The ecologist's field guide to sequence-based identification of biodiversity. Methods in Ecology and Evolution 7(9): 1008-1018.


Deiner, K., Bik, H.M., Mächler, E., Seymour, M., Lacoursière-Roussel, A., Altermatt, F., Creer, S., Bista, I., Lodge, D.M. and De Vere, N. 2017. Environmental DNA metabarcoding: Transforming how we survey animal and plant communities. Molecular Ecology 26(21): 5872-5895.



Dormontt, E.E., Boner, M., Braun, B., Breulmann, G., Degen, B., Espinoza, E., Gardner, S., Guillery, P., Hermanson, J.C. and Koch, G. 2015. Forensic timber identification: It's time to integrate disciplines to combat illegal logging. Biological Conservation 191: 790-798.


Gasson, P. 2011. How precise can wood identification be? wood anatomy’s role in support of the legal timber trade, especially CITES. IAWA Journal 32(2): 137-154.


Gernandt, D.S., López, G.G., García, S.O. and Liston, A. 2005. Phylogeny and classification of
pinus. Taxon 54(1): 29-42.


Goryachkina, O.V., Badaeva, E.D., Muratova, E.N. and Zelenin, A.V. 2013. Molecular cytogenetic analysis of siberian
larix species by fluorescence in situ hybridization. Plant Systematics and Evolution 299: 471-479.


Hubert, F., Grimm, G.W., Jousselin, E., Berry, V., Franc, A. and Kremer, A. 2014. Multiple nuclear genes stabilize the phylogenetic backbone of the genus
quercus. Systematics and Biodiversity 12(4): 405-423.


Jiao, L., Lu, Y., He, T., Guo, J. and Yin, Y. 2020. DNA barcoding for wood identification: Global review of the last decade and future perspective. IAWA Journal 41(4): 620-643.


Nakase, T., Tsuzuki, S., Lee, F., Sugita, T., Jindamorakot, S., Jan-ngam, H., Potacharoen, W., Tanticharoen, M., Kudo, T. and Takashima, M. 2003.
Sporobolomyces magnisporus sp. nov., a new yeast species in the
erythrobasidium cluster isolated from plants in Taiwan. The Journal of General and Applied Microbiology 49(6): 337-344.



Nellemann, C. 2012. Green carbon, black trade: Illegal logging, tax fraud and laundering in the world's tropical forests.

Reboredo, F. 2013. Socio-economic, environmental, and governance impacts of illegal logging. Environment Systems and Decisions 33: 295-304.


Zeb, U., Dong, W., Zhang, T., Wang, R., Shahzad, K., Ma, X. and Li, Z. 2020. Comparative plastid genomics of
pinus species: Insights into sequence variations and phylogenetic relationships. Journal of Systematics and Evolution 58(2): 118-132.


Zhou, B.F., Yuan, S., Crowl, A.A., Liang, Y.Y., Shi, Y., Chen, X.Y., An, Q..Q., Kang, M., Manos, P.S. and Wang, B. 2022. Phylogenomic Analyses Highlight Innovation and Introgression in the Continental Radiations of Fagaceae Across the Northern Hemisphere. Nature Communications 13(1): 1320